BioDivErSI mira a realizzare una strumentazione prototipo versatile, che usi una sorgente a “electrospray ionization”, ESI, connessa ad uno strumento per spettrometria di massa [2] e altre fonti di ionizzazione per 1) una efficiente selezione delle molecole delle specie biologiche presenti nel campione in analisi e 2) una loro caratterizzazione in termini di m/z. Questa analisi, combinata con predizioni di DM e ML, permetterà di risalire alla composizione della sequenza nucleotidica del miRNA e alla struttura secondaria che assume durante l’interazione con le molecole target. L’obiettivo verrà perseguito con la costruzione, caratterizzazione e ottimizzazione di uno strumento
prototipo attraverso fasi successive, ciascuna con propri obiettivi e risultati, che permetteranno di valutare lo stato di avanzamento del progetto e hanno di per sé una propria valenza scientifica.
Essendo i miRNA i biomarker rilevanti nel progetto, la loro identificazione rappresenterà il “fil rouge” per lo sviluppo del medesimo attraverso l’analisi di sequenze modello, la costruzione di un data-base e l’analisi computazionale e sperimentale dei complessi miRNA-molecola target.
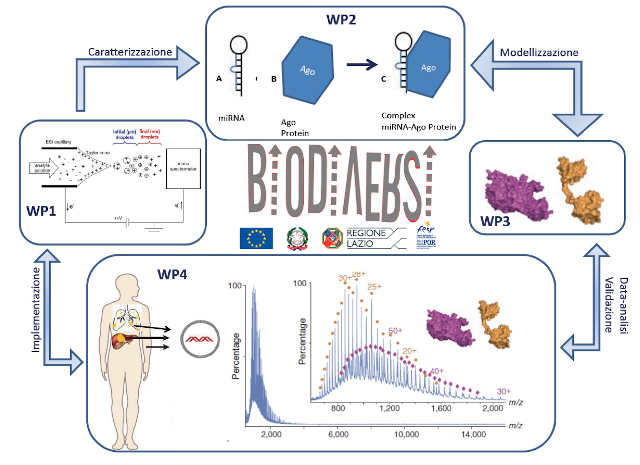
Il progetto sarà implementato con quattro workpackage (WP). WP1: Realizzazione ed ottimizzazione del sistema di electrospray ionization accoppiato al sistema di spettroscopia di massa (ESI-MS). WP2: Caratterizzazione dei miRNA via ESI-MS, come biomolecole singole e in complessi con mRNA target. WP3: Studio computazionale della struttura dei miRNA isolati ed in complessi con tecniche di DM e ML. WP4: Validazione della metodologia sviluppata su sistemi prototipo, verifica di un possibile trasferimento del know-how e diffusione dei risultati. Le interazioni tra i WP sono descritte nel diagramma Pert, mentre la programmazione dei WP è presentata nel diagramma di Gantt con le milestone e i deliverable.
 |
 |

