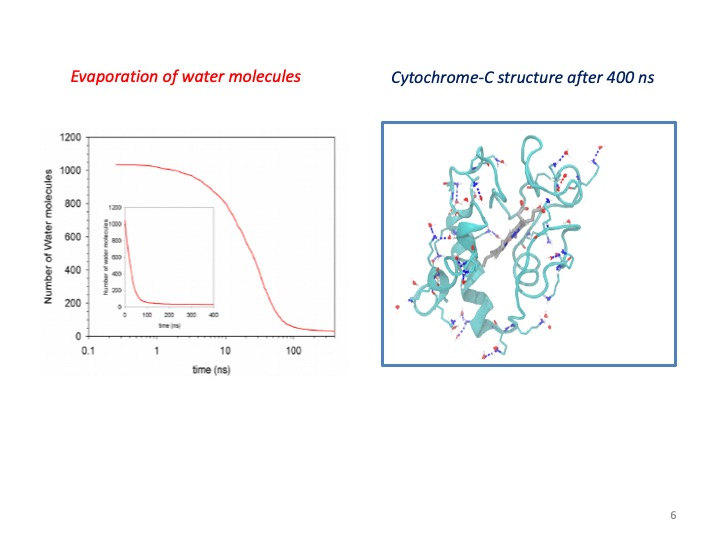
WP2 : Studio computazionale dell’interazione enzima-solvente ed enzima-supporto. Responsabile : S. Borocci (Università della Tuscia). Partecipanti : A. Casavola (CNR-ISM) e A. Cartoni (Sapienza Università di Roma)
Le finalità di questo WP sono - mettere a punto una metodologia teorica computazionale predittiva per lo studio dell’ interazione enzima -solvente - indirizzare la scelta delle coppie enzima/supporto più promettenti con particolare attenzione ai materiali valutati idonei per lo sviluppo di biosensori. Il WP si articola in 3 attività: Attività 2.1 Scelta e sviluppo delle metodologie computazionali Attività 2.2 Studio dell’ interazione enzima-solvente e confronto della struttura molecolare dell’enzima in fase liquida e gassosa Attività 2.3 Studio dell’ interfaccia enzima/supporto nei sistemi di interesse del progetto. Per questa attività è stata acquisita una workstation dotata di un sistema multiprocessore con scheda grafica GPU per calcolo parallelo intensivo secondo lo schema ibrido CPU/GPU (WorkstaNon HP Z6, CPU:1 CPU da 96GB(3x32GB); GPU :NVIDIA Quadro P5000 16GB). La workstation è stata istallata presso il Dipartimento per l’Innovazione dei sistemi biologici, agroalimentari e forestali dell’Università della Tuscia, Viterbo, e messa in rete per poter essere utilizzata sia dal gruppo del CNR-ISM a Montelibretti che dalla dott.sa Cartoni del Dipartimento di Chimica, Sapienza Università di Roma. Il server è completamente dedicato alle simulazioni di dinamica molecolare ed ai calcoli della struttura di enzimi e proteine. Per le simulazione di dinamica molecolare si è scelto di utilizzare il software open-source GROMACS, con licenza gratuita LGPL (Lesser General Public License).